作者:汪晶晶
光鑷——一種利用光捕獲與操控微小物體的工具。2018年,這項 “爆冷”的技術奪得諾貝爾物理學獎的桂冠,也終于實現了“光捕獲之父” Arthur Ashkin的預言:“我想生物學領域可能會因為用光鑷做出的偉大工作而獲得一次諾貝爾獎。”本期將從光鑷在生物學領域的應用發展方面做一概述。
一、光鑷的由來
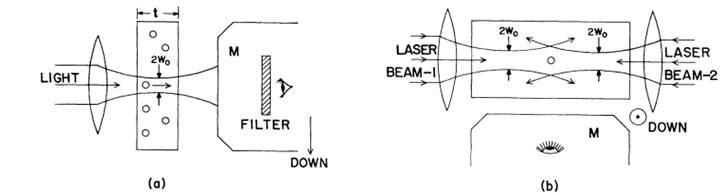
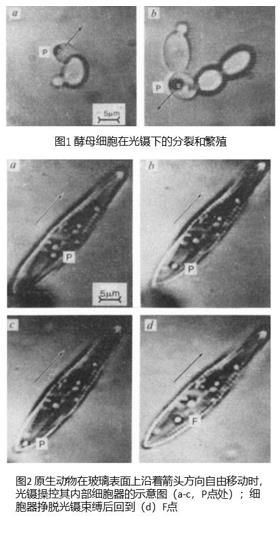
光鑷技術的核心是光與物質之間的相互作用。在這種相互作用中產生的微力可以用來移動和捕獲微小物體。1970年,Ashkin奠定了現代光鑷技術的基礎。在Bell實驗室,Ashkin發現微米尺寸的乳膠球(beads)可以被幾毫瓦的氬離子激光束吸引到中央(Ashkin 1970)(圖a),就是這種引力讓光捕獲得以實現。Ashikin也觀察到,激光散射驅使小球移動。通過使用兩束相反的激光,他成功避免了前驅力,最終創造了使小球懸浮在水中的首個穩定光阱(圖b)。
1986年,Ashkin和Chu等提出了光鑷的現在形式——使用一束緊密聚集的激光,在三維空間中穩定地捕獲直徑在25nm到10μm之間的微粒。(Ashkin, Dziedzic et al. 1986;Lin, Fujita et al.2017)(圖c)。
二、光鑷在生物學領域的應用與發展
光鑷在生物學領域有廣泛應用。一個重要的原因是它為生物學家提供了一只“微觀手”去操控生物體,像光學顯微鏡技術一樣具有低侵入性。另外,光鑷可獲得的長度、時間和力學尺度在生物學上從單細胞到單分子水平是具有相關性的。
1987年,Ashkin發表了光鑷在生物學中應用的第一篇文章——操控單個病毒和活細菌(Ashkin and Dziedzic 1987)。通過選擇合適的激光功率和波長,使生物樣品的光毒性最小化,以捕獲和操控單個活細胞(Ashkin, Dziedzic et al. 1987)。
90年代初期,光鑷的使用已擴展到單個生物分子水平。在這些單分子研究中,感興趣的生物分子本身并不是直接被捕獲的,而是被光學捕獲的微球所操縱,這些微球起著手柄和力傳感器的作用(圖3)。這種單分子研究的很大一部分包括對單個驅動蛋白活動的研究(Svoboda,Schmidt et al. 1993;Yin,Wang et al. 1995)。這些蛋白移動產生的力和運動可以通過光鑷進行研究和控制,用以揭示它們的動力學和能量學特性。
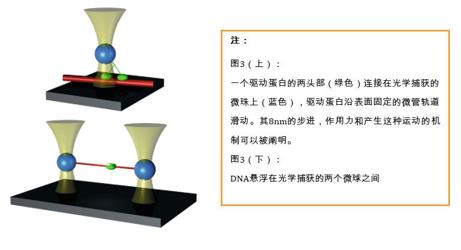
圖3
另一個重要的研究領域是DNA。在這些研究中,DNA分子粘附于一個或多個光捕獲的小球上,這使得分子可以被拉伸,并通過力學譜研究它們的機械特性(Smith,Cui et al. 1996;Bustamante,Bryant et al. 2003)(圖4)。
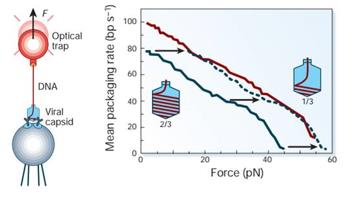 圖4 噬菌體DNA的組裝
圖4 噬菌體DNA的組裝
當DNA被噬菌體?29馬達拉入以填充病毒衣殼時,外部DNA的延伸變短。當衣殼被填充三分之二(藍色實線)時,組裝的力-速度曲線必須偏移約15pN(藍色虛線),才與衣殼被填充三分之一(紅色)時的曲線相符。提示在DNA組裝過程中存在因DNA高度壓縮而形成的其他內力(將DNA注入宿主細胞的驅動力)。
另外,這些設計被用來研究蛋白與DNA蛋白之間的相互作用(Davenport,Wuite et al. 2000;Dame,Noom et al. 2006;Neupane,Foster et al. 2016)。許多DNA與蛋白之間的相互作用影響DNA的結構,從而影響DNA分子的長度(力學依賴性)。利用光鑷,這些長度變化可以通過測量微球的位移來觀察(圖5)。例如DNA-結合蛋白以及DNA和RNA聚合酶活性的研究。
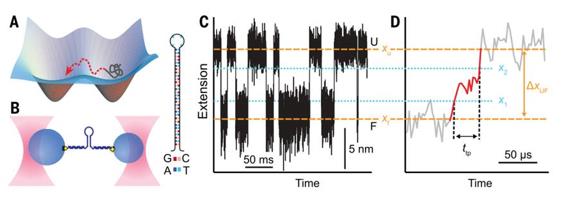 圖5 過渡態路徑的力學譜測量
圖5 過渡態路徑的力學譜測量
A. 過渡態路徑(transition paths)代表蛋白質折疊過程中跨越能量障礙的短暫過程(紅線);與之形成對比的是大部分時間在勢阱中波動的過程(灰色線)
B. DNA發夾連接在兩段“手鏈”結構上,分別附著在光捕獲的兩個微球上
C. 恒力狀態下,發夾結構在折疊和去折疊狀態間維持著動態變化
D.過渡路徑(紅線):在U(unfolding)和F(folding)之間移動的軌跡
在過去十年中,光鑷與其他技術的結合進一步擴大了光鑷在生物學領域的影響。包括與多通道微流控系統的結合(Brewer andBianco 2008),不僅提高了實驗通量,還提供了更復雜的多步生物學反應過程的原位控制。最引人注目的是,光鑷與豐富多樣的熒光技術相結合,使得光鑷的分析范疇遠遠超過了機械測量(Heller, Sitterset al. 2013;Block, Witt etal. 2017)(圖6)。
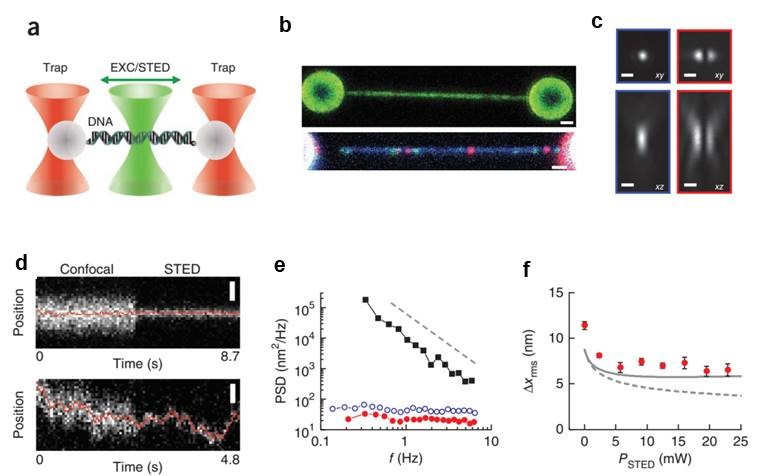
圖6 光鑷與共聚焦和STED熒光顯微鏡相結合,可視化單個DNA結合蛋白在DNA上的移動軌跡
a. DNA兩端連在兩個光鑷捕獲的微球之間,在DNA上掃描的激發光束(EXC)和疊加的STED光束顯示為綠色。b. λDNA和3.2-μm微球的共聚焦熒光圖像。c. 共聚焦和1D STED激光束示意圖(左側:共聚焦圖像;右側:STED圖像;scale bar:500nm)。d. 光學拉伸DNA上的Atto 647n標記蛋白波動曲線(部分共聚焦,部分STED)。e. 功率譜密度(PSD): 跟蹤蛋白質在DNA上的位置。f. 定位精度?Xrms 與PSTED的函數關系圖。
隨著近年來商業化光鑷系統的出現,這種強大的單分子技術正趨于成熟,為越來越多的生物科學家所使用。隨著商業化熒光技術和原子力顯微鏡(AFM)技術的發展,可以預期商業化光鑷將極大地促進我們在單分子水平上的生物學認識。
諾貝爾獎得主朱棣文(Steven Chu)曾在《Physics Today》中說過,“如果在未來十年,另一個諾貝爾獎得主是源于他通過光鑷或其他單分子技術在分子生物學領域有了突破性發現,他不會感到驚訝。”(文末附參考文獻)
本期參考文獻
1. Ashkin,
A. (1970)."Acceleration and Trapping of Particles by Radiation
Pressure." PhysicalReview Letters 24(4): 156-159.
2. Ashkin,
A. and J. M. Dziedzic (1987). "Optical trapping and manipulation of
viruses and bacteria." Science(New York, N.Y.) 235(4795):1517-1520.
3. Ashkin,
A., J. M. Dziedzic, J. E.Bjorkholm and S. Chu (1986). "Observation of a
single-beam gradient forceoptical trap for dielectric particles." Opt Lett
11(5): 288.
4. Ashkin,
A., J. M. Dziedzic and T.Yamane (1987). "Optical trapping and manipulation
of single cells usinginfrared laser beams." Nature 330(6150):769-771.
5. Block,
J., H. Witt, A. Candelli,E. J. G. Peterman, G. J. L. Wuite, A. Janshoff and S.
K?ster (2017)."Nonlinear Loading-Rate-Dependent Force Response of Individual
VimentinIntermediate Filaments to Applied Strain." Physical review letters
118(4): 048101-048101.
6. Brewer,
L. R. and P. R. Bianco(2008). "Laminar flow cells for single-molecule
studies of DNA-proteininteractions." Nature methods 5(6):517-525.
7. Bustamante,
C., Z. Bryant and S.B. Smith (2003). "Ten years of tension:
single-molecule DNAmechanics." Nature 421(6921):423-427.
8. Chu, S.
(1991). "Lasermanipulation of atoms and particles." Science
253(5022): 861-866.
9. Dame, R.
T., M. C. Noom and G. J.L. Wuite (2006). "Bacterial chromatin organization
by H-NS proteinunravelled using dual DNA manipulation." Nature 444(7117):
387-390.
10. Davenport,
R. J., G. J. Wuite, R.Landick and C. Bustamante (2000). "Single-molecule
study oftranscriptional pausing and arrest by E. coli RNA polymerase."
Science(New York, N.Y.) 287(5462):2497-2500.
11. Heller,
I., G. Sitters, O. D.Broekmans, G. Farge, C. Menges, W. Wende, S. W. Hell, E.
J. G. Peterman and G.J. L. Wuite (2013). "STED nanoscopy combined with
optical tweezers revealsprotein dynamics on densely covered DNA." Nature
methods 10(9): 910-916.
12. Lin, H.
Y., N. Fujita, K. Endo,M. Morita, T. Takeda, T. Nakagawa and R. Nishimura
(2017). "Isolation andCharacterization of Multipotent Mesenchymal Stem
Cells Adhering to Adipocytesin Canine Bone Marrow." Stem Cells Dev 26(6):
431-440.
13. Neupane,
K., D. A. N. Foster, D.R. Dee, H. Yu, F. Wang and M. T. Woodside (2016).
"Direct observation oftransition paths during the folding of proteins and
nucleic acids." Science(New York, N.Y.) 352(6282):239-242.
14. Smith,
S. B., Y. Cui and C.Bustamante (1996). "Overstretching B-DNA: the elastic
response ofindividual double-stranded and single-stranded DNA molecules."
Science(New York, N.Y.) 271(5250):795-799.
15. Svoboda,
K., C. F. Schmidt, B. J.Schnapp and S. M. Block (1993). "Direct
observation of kinesin stepping byoptical trapping interferometry." Nature
365(6448): 721-727.
16. Yin, H.,
M. D. Wang, K. Svoboda,R. Landick, S. M. Block and J. Gelles (1995).
"Transcription against anapplied force." Science (New York, N.Y.)
270(5242): 1653-1657.
本文章版權歸清華大學生物醫學測試中心細胞生物學平臺所有